La trisomie 21 est un syndrome polymalformatif avec un retard mental, lié dans la majorité des cas à la présence d'un chromosome 21 surnuméraire.
En France, le dépistage de la trisomie 21 est proposé à toutes les femmes enceintes au 1er trimestre de grossesse. Ce dépistage consiste à calculer un score en fonction de marqueurs sanguins (AFP, Papp-A, beta HCG) et des signes d'appel échographique comme la clarté nuccale. Si ce score dépasse un certain seuil, un diagnostic cytogénétique est proposé pour confirmer la trisomie. Il consiste à prélever des cellules fœtales par des techniques invasives comme l'amniocentèse et de dénombrer le nombre de chromosomes sur un caryotype comme illustré ci-dessous.
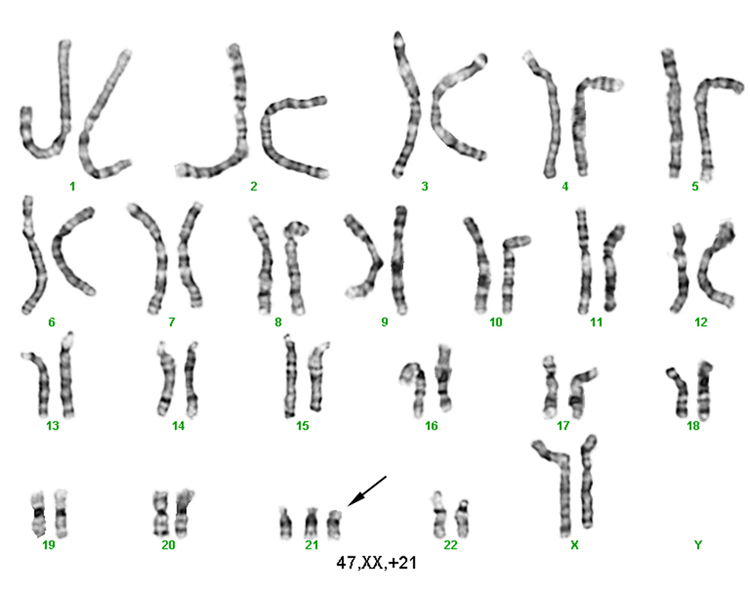
Malheureusement ces gestes invasifs ne sont pas anodins. On estime entre 0.5% et 1% le risque de fausse couche lié à l'amniocentèse.
Cela peut sembler faible, mais le dépistage actuel souffre d'une très mauvaise spécificité. C'est à dire que beaucoup de femmes répondent positif au dépistage alors que leur fœtus est indemne. Par conséquence beaucoup trop d'amniocentèses sont réalisées inutilement avec le risque de fausse couche qui en découle.
Mais depuis l’avènement récent du séquençage haut débit, un nouveau test de dépistage beaucoup plus puissant en terme de sensibilité et de spécificité voit le jour. Il s'agit du DPNI pour Dépistage Prénatal Non Invasif. (Vous lirez souvent diagnostic , mais il s'agit pour l'heure de dépistage). Ce nouveau test consiste à quantifier sur une simple prise de sang un excès d'ADN fœtal circulant provenant du chromosome 21.
L'ADN fœtal circulant
Il y a des fragments d'ADN double brin qui circulent librement dans votre sang. En général ce sont les vôtres sauf si vous faites des expériences tordues dans votre laboratoire ou ... si vous êtes enceinte. En effet chez les femmes enceintes environ 10% de ces fragments proviennent du fœtus. Plus précisément ces fragments proviennent de la lyse des cellules trophoblastiques, un composant du placenta ayant la même origine embryologique que le fœtus.
L'idée derrière le DPNI c'est de quantifier l' excès d'ADN circulant provenant du chromosome 21 à l'aide des nouvelles technologies de séquençage haut débit.
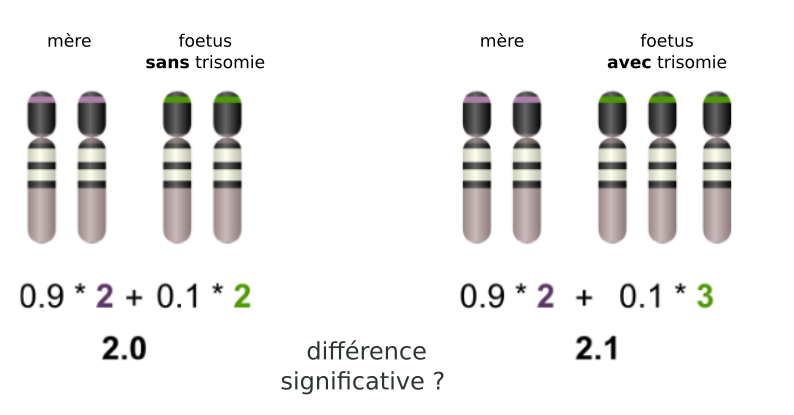
La figure ci-dessous illustre la quantification d'ADN circulant chez une mère sans et avec fœtus trisomique. En mesurant une différence significative entre une patient et des témoins sains, il est possible de conclure à un excès de d'ADN circulant provenant du chromosome 21.
Le Séquençage de nouvelle génération
Le NGS (Next Generation Sequencing) est une technologie récente permettant le séquençage de l'ADN de façon très rapide grâce à un haut niveau de parallèlisation. Sans entrer dans les détails (super site ici), les fragments d'ADN provenant du sang maternel, qu'on appellera reads à présent, sont séquencés puis alignés sur le génome de référence via des algorithmes de bioinformatique.
On obtient alors un fichier contenant la liste des reads associés à leurs positions sur le génome. C'est à dire qu'à chaque fragment séquencé, son chromosome lui est associé.
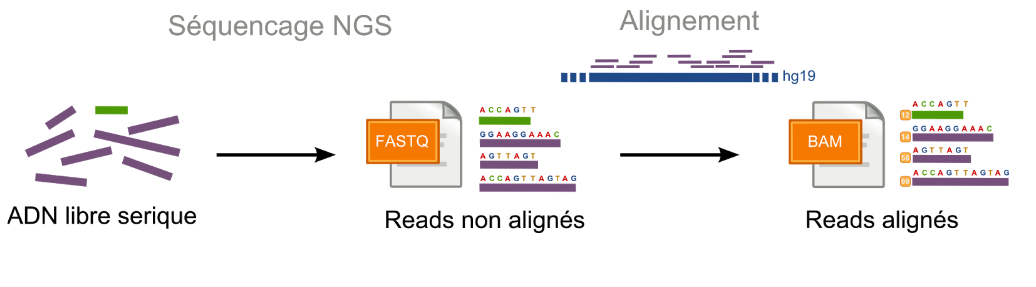
La figure ci-dessous résume les étapes du séquençage ainsi que les différents formats de fichier.
Quantification et test statistique
Une trisomie 21 se caractérise par un excès de reads s’alignant sur le chromosome 21. Pour mesurer cet excès il nous faut des valeurs de référence obtenues chez des femmes enceintes témoins dont le fœtus est sain.
Avec un nombre suffisant de témoins, la moyenne et l'écart-type du nombre de reads par chromosome sont calculés.
Pour savoir si une patiente présente trop de reads, il suffit de rechercher une différence significative à l'aide d'un Z-score en comparant les données de la patiente et les valeurs de référence.
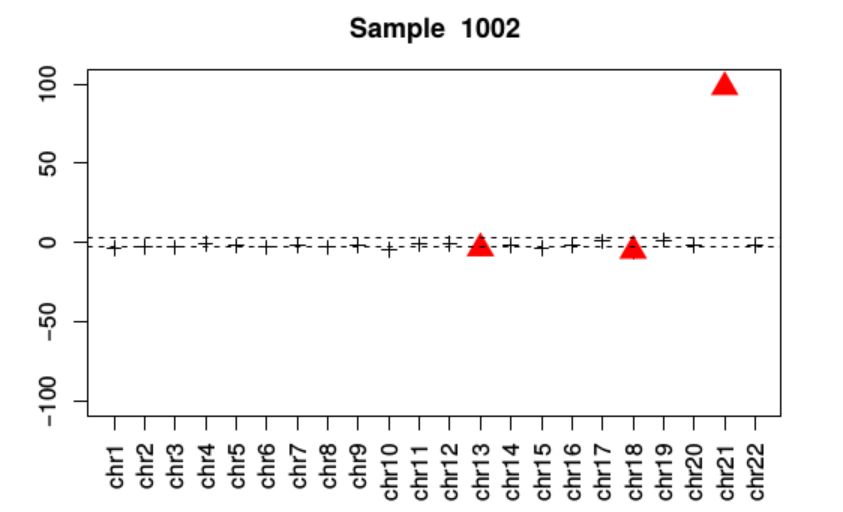
Le logiciel RapidR créé par le NHS utilise cette approche. Un exemple de résultats est présenté dans la figure suivante.
Un autre logiciel, Wisecondor approche différemment le problème et propose d'utiliser les autres chromosomes comme références au sein du même échantillon. C'est une approche plus complexe, mais peut se résumer ainsi.
Tout d'abord, le génome est segmenté en régions de 10 kilobases appelé bin. Le nombre de reads est comptabilisé par bin au lieu d'être comptabilisé par chromosome comme précédemment.
A partir des mesures chez les témoins sains, les bins du chromosome 21 sont associés aux autres bins du génome lorsqu'ils contiennent plus ou moins le même nombre de reads. La figure suivante est une représentation de ces associations.
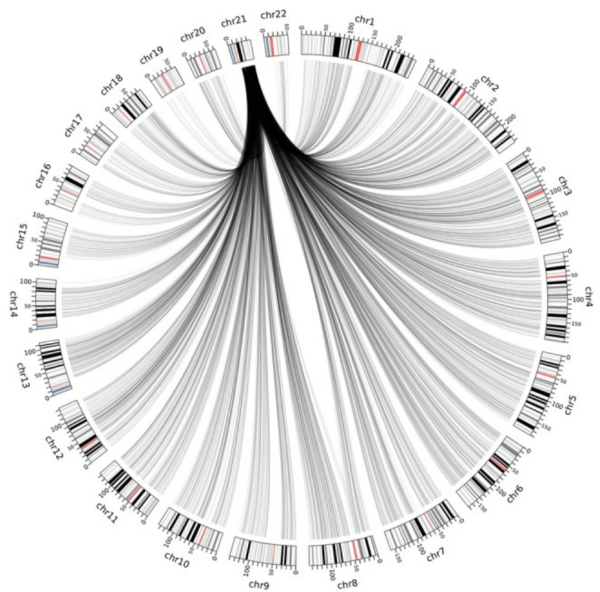
Ce logiciel permet donc de mesurer l'excès de reads mappant le chromosome 21 en le comparant aux autres chromosomes. Cette technique à l'avantage de se passer des biais de mesure, car le séquençage de la référence et du patient sont réalisés en même temps.
Stratégie de dépistage
En Novembre 2015, la Haute autorité de santé a évalué positivement ce nouveau test de dépistage. La sensibilité et la spécificité avoisine les 100%.
Le risque de faux négatif est lié à l'absence d'ADN fœtal si le prélèvement est réalisé trop tôt. Un risque de faux positif est également possible dans les cas de mosaïcisme fœtal.
Aujourd'hui toutes les femmes enceintes, quel que soit leur âge, sont informées de la possibilité de recourir à ce test.
La stratégie actuelle est de proposer le dépistage standard. Si celui ci revient positif, au lieu de proposer directement un geste invasif, le DPNI est proposé. Si celui ci est à son tour positif, alors le geste invasif et le diagnostic cytogénétique est réalisé.
A l'heure actuelle les laboratoire Cerba et Biomnis proposent le DPNI. Mais depuis janvier 2016 certains centres hospitaliers proposent ce test qui n'est malheureusement pas encore remboursé et avoisine les 800€.
Référence
- Stratégies de dépistage de la trisomie 21
- Les performances des tests ADN libre circulant
- La valeur d'un dépistage préatal non invasif ( Illumina)
- RapidR
- Wisecondor
Remerciement
Ce site est versionné sur GitHub. Vous pouvez corriger des erreurs en vous rendant à cette adresse
Go Top