Envie d'identifier le criminel qui vous a volé au boulot votre bic 4 couleurs en votre absence ? Dans ce cas, cet article est fait pour vous ! Dans ce billet nous allons voir comment, à l'aide des séquences répétées dans le génome humain, il est possible d'identifier une personne en lui attribuant un « code-barres génétique ».
On se met tout de suite la musique des experts Manhatthan et on commence !
Les séquences répétées
10 % du génome humain est constitué de séquences d'ADN répétées en tandem. Il s'agit de séquences plus ou moins longues, appelées « noyaux », « motifs » ou encore « unités de répétition » (ex : GAAA), et se répètent successivement un certain nombre de fois (ex : GAAAGAAAGAAAGAAAGAAA).
Ces séquences répétées sont présentes partout dans le génome, principalement dans les télomères et les centromères. Parfois, ces séquences se trouvent à proximité de gènes codants et une modification du nombre de répétitions peut alors entraîner des répercussions cliniques. L'exemple type est la maladie de Huntington. Cette atteinte neurodégénérative héréditaire est caractérisée par une expansion de triplets CAG supérieure à 30 dans le gène HTT de l'huntingtine.
On distingue 2 types de séquences répétées en fonction de la taille du motif. Les minisatellites ou VNTR (Variable Number Tandem Repeat) contiennent un motif de 9 à 80 bases et les microsatellites ou STR (Short Tandem Repeat) un motif de 2 à 5 bases. Ce sont ces dernières qui sont utilisées pour l'identification des personnes par empreinte génétique.
Le polymorphisme
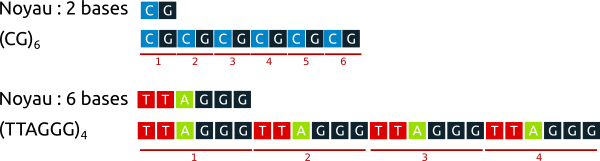
La variation du nombre de répétitions varie fortement dans la population. Par exemple, pour une position génomique donnée, un individu (bleu) pourrait avoir sur son chromosome paternel la répétition (CG)6 et sur son chromosome maternel la répétition (CG)8. Un autre individu (rouge) pourrait porter sur ses chromosomes les allèles (CG)6 et (CG)9.

L'identification de plusieurs régions répétées au sein du génome permet d'associer à un individu une combinaison unique. Une palette de 13 loci + 2 loci (oui, un locus... des loci) sur les chromosomes sexuels est aujourd'hui utilisée par la police scientifique pour identifier n'importe quel individu. Le caryotype ci-dessous montre la position et le nom de ces STR sur les chromosomes.
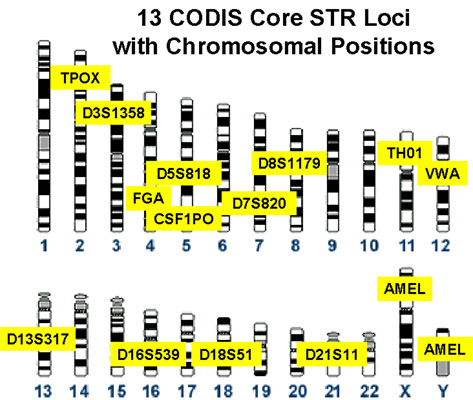
Identification des STR
Pour créer une empreinte génétique, il suffit tout simplement de mesurer la taille de ces 13 régions répétées en les amplifiant par PCR. Pour cela, pour chaque STR, on utilise un couple d'amorces flanquant le STR en question. Une des deux amorces est couplée à un fluorochrome qui permet ensuite l'identification de la séquence par électrophorèse capillaire. Les séquences des amorces sont disponibles ici.

À la fin de la PCR, on obtient des amplicons dont la taille est proportionnelle à celle du STR. Une analyse de fragments est ensuite réalisée à l'aide d'un séquenceur capillaire. En d'autres termes, les amplicons migrent dans un capillaire plus ou moins vite et leur temps de passage est mesuré lors de la détection du fluorochrome par un laser. Les résultats sont représentés par des pics de fluorescence dont la position sur l'axe des abscisses correspond à la taille du STR.
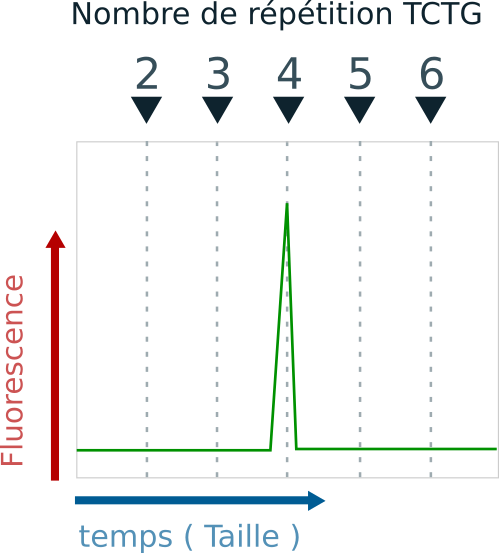
Prenons par exemple un individu homozygote pour le locus vWA. À ce locus, cet individu possède 4 répétitions TCTG à la fois sur le chromosome maternel et sur le chromosome paternel. Son génotype pourrait s'écrire : (TCTG)4 / (TCTG)4. Dans ce cas, la PCR amplifie des amplicons tous de la même taille et un seul pic est détecté avec l'analyse de fragments.
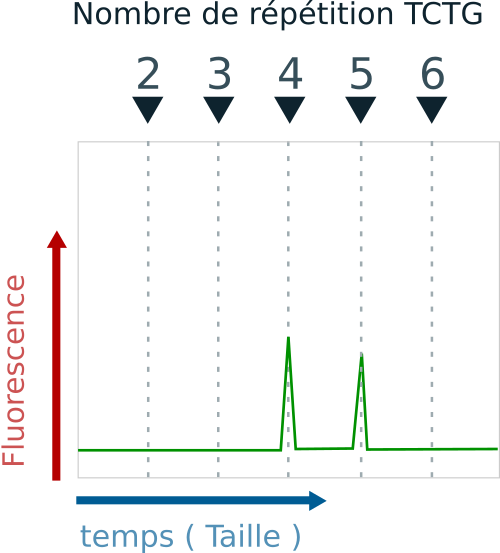
En revanche, si le patient est hétérozygote avec le génotype suivant : (TCTG)4 / (TCTG)5, on observe 2 pics et une diminution des amplitudes.
Pour créer une empreinte génétique, il suffit de refaire la même chose pour les 13 loci. On fait une PCR multiplexe en mélangeant tous les couples d'amorces et en discriminant chaque locus à l'aide de 4 fluorochromes différents ainsi que par des tailles de STR différentes. En traçant l'ensemble des pics, on obtient l'empreinte génétique. La probabilité que deux individus (non jumeaux) aient le même profil est extrêmement faible, de l'ordre de 10-10.
Voici le profil d'un individu que j'ai trouvé sur Google !
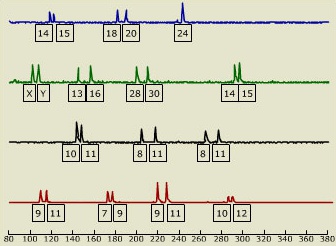
On retrouve nos 13 loci ainsi que 2 loci sur les chromosomes sexuels XY, qui nous informent sur le sexe. Sur la première ligne en bleu, on peut observer 3 loci (TPOX, FGA et vWA). L'individu est hétérozygote pour le premier locus avec 2 allèles présentant 14 et 15 répétitions respectivement ; puis un deuxième locus également hétérozygote (18/20) et enfin un troisième locus, cette fois homozygote avec 24 répétitions.
Vous pouvez refaire la même chose pour les autres loci et avoir le profil complet de cette empreinte génétique.
Notons également, que chaque allèle observé provient d'un des parents. Par exemple pour le premier locus (14/15), l'allèle 14 peut provenir de la mère et l'allèle 15 du père. En recherchant les empreintes génétiques chez les parents, nous pourrions confirmer le lien de filiation.
Conclusion
Voilà, vous savez faire une empreinte génétique. Vous allez maintenant pouvoir la comparer avec une banque de données. C'est aux États-Unis en 1994 que la première banque de données d'empreintes génétiques a été créée sous l'égide du FBI, sous le label CODIS. En France, suite à l'affaire du tueur parisien Guy Georges, la loi du 17 juin 1998 acte la création du fichier national (FNAEG). Il recense aujourd'hui 2 655 381 personnes.
Références
- Brief Introduction to STRs
- Les kits commerciaux
- Liste des amorces
- The rarity of DNA profiles
- Pour mieux comprendre l'analyse de fragments
Remerciement
- @Piplopp
- @Oodnadatta
Ce site est versionné sur GitHub. Vous pouvez corriger des erreurs en vous rendant à cette adresse
Go Top