MyHeritage, 23andMe, Ancestry… Vous en avez sûrement entendu parler. Bien qu'interdits en France (article 16-10), ces tests génétiques en libre accès bénéficient pourtant d'une bonne publicité. Depuis peu, elle se fait par l'intermédiaire de YouTube. Amixem, Squeezie et plus récemment Dr Nozman ont fait ces tests, puis ont partagé leurs résultats en vidéo. Devant cet engouement, j'ai voulu savoir quelles informations étaient rendues à l'utilisateur et si des diagnostics médicaux pouvait être établis d'après les résultats.

La génétique à deux vitesses
Avant tout, j'aimerais vous montrer le paradoxe de ces tests génétiques, qu'ils soient réalisés dans un cadre médical ou directement sur Internet.
En médecine, ces tests sont très probablement les analyses les plus contrôlées de toutes les analyses de biologie médicale. Ils sont sous l'égide de l'Agence de la biomédecine et sont prescrits uniquement par des médecins généticiens ou par des médecins non généticiens, mais travaillant en relation avec une équipe de génétique clinique. Un consentement (exemple) est obligatoirement demandé aux patients. En oncogénétique, les analyses sont souvent réalisées deux fois pour éviter une erreur sur l'identité. Pour signer des résultats, même après mon doctorat de médecine, j'ai dû attendre d'avoir les agréments de l'Agence de la biomédecine…
Et en même temps, il n'a jamais été aussi facile de faire un test génétique sur Internet. Le fait de ne pas avoir besoin de faire une prise de sang les rend même plus faciles et plus accessibles qu'un examen classique de biochimie ou d'hématologie. On clique sur un bouton, on reçoit un kit salivaire par la poste et le tour est joué.
Mais alors, pourquoi un tel contrôle en médecine ?
D'abord, à cause du caractère de la génétique à pouvoir prédire des maladies chez des personnes en bonne santé. Contrairement à un examen classique qui teste des patients malades, les examens de génétique concernent souvent des individus sains. C'est une médecine aussi bien de diagnostic (identifier une maladie chez un patient) que de prédiction (identifier une maladie susceptible de se déclarer). Par exemple, retirer les seins d'une femme en bonne santé parce qu'elle est porteuse d'une mutation prédisposant au cancer reste une décision délicate à prendre.
Ensuite, il y a l'hérédité. Vos gènes ne vous appartiennent pas, vous les partagez avec votre famille. Faire votre propre diagnostic implique aussi de le faire chez les membres de votre famille. Et donc si vous réalisez ce type de test génétique, les résultats peuvent vous déclarer porteur d'une mutation, et indirectement suggérer que vos parents, vos frères, vos sœurs, vos enfants partagent également cette mutation. Cela n'impliquera donc pas que vous.
Un test de généalogie qui en dit plus ?
La plupart de ces tests sont vendus comme une analyse sur vos origines ethniques. La société MyHeritage utilise pour cela une puce à ADN permettant d'identifier environ 700 000 variations génétiques appelées SNV (Single Nucleotide Variant) absentes ou présentes de votre génome, qu'elle compare à des populations de référence. Une des méthodes est d'identifier des groupes de SNV (haplogroupes) permettant d'associer un individu à sa population d'origine. Mais alors, qu'est-ce que ces SNV et que peuvent-ils dire de plus ?
Single Nucleotide Variant (SNV)
Un génome humain est constitué de 3 milliards de bases, représentées par les lettres A, C, G et T. Elles sont réparties sur 46 chromosomes différents (22 paires de chromosomes de 1 à 21 + 1 paire XX ou XY selon que vous êtes une femme ou un homme, respectivement). Chacune de vos cellules est constituée de 2 versions de ce génome, héritées de votre mère et de votre père.
Un SNV est une modification d'une lettre d'un génome (le vôtre par exemple) par rapport à un génome de référence (séquence d'ADN assemblée par les scientifiques, représentative d'une espèce — ici l'humain – réalisée à partir de plusieurs personnes). Si par exemple, à la position 101594229 du chromosome 9 on vous identifie un A, mais sur le génome de référence il y a un G, alors vous avez un SNV qui peut s'écrire : chr9:101594229G>A. Une partie de ces variations observées sont référencées dans des bases de données de variations génétiques, comme la base de données dbSNP qui attribue à des SNV fréquents un identifiant unique (rs145236923).
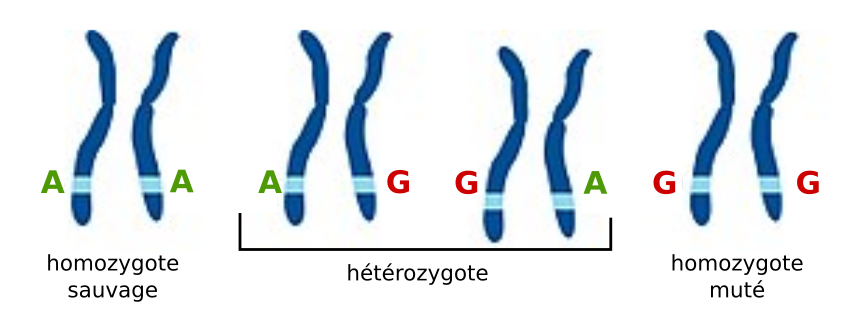
Quant au génotype d'un SNV, il indique si vous portez la variation sur un seul ou sur les deux chromosomes hérités de vos parents. Pour une variation donnée, il y a trois génotypes possibles : homozygote sauvage, hétérozygote (sauvage et muté) et homozygote muté.
- GG : vous n'êtes pas porteur du SNV (sur aucun chromosome), vous êtes homozygote sauvage ;
- AG : vous portez le SNV à l'état hétérozygote (sur un seul chromosome) ;
- AA : vous portez le SNV à l'état homozygote (sur deux chromosomes), vous êtes homozygote muté.
Les puces à ADN permettent d'identifier les génotypes de milliers de SNV préalablement choisis. Le fichier de résultat brut fourni par MyHeritage donne l'identifiant du SNV, sa localisation (chromosome, position) et le génotype pour le p̶a̶t̶i̶e̶n̶t̶ client :
# Exemple de fichier brut
rs28678693 1 838665 CC
rs4475691 1 846808 CT
rs72631889 1 851390 TT
Comment interpréter ces SNV ?
Sur les 3 milliards de bases du génome, il y a chez un individu environs 1 SNV toutes les 1000 bases qui le distingue d'un autre individu. La majorité d'entre elles sont bénignes, mais certains peuvent être pathogènes. En génétique médicale, on classe ces variations en 5 classes différentes :
- Classe 1 : variation bénigne (benign)
- Classe 2 : variation probablement bénigne (likely benign)
- Classe 3 : variation de signification indéterminée (unknown significance)
- Classe 4 : variation probablement pathogène (likely pathogenic)
- Classe 5 : variation pathogène (pathogenic)
Cette classification se fait à l'aide d'arguments scientifiques plus ou moins forts, résumés dans ce qu'on appelle les recommandations de l'ACMG. Par exemple, on peut se poser certaines questions.
- La variation est-elle déjà décrite dans la littérature (déjà connue) ?
- La variation est-elle rare ou fréquente dans la population (beaucoup d'autres personnes l'ont aussi) ?
- Est-elle située dans un gène ou non (située dans les régions codantes du génome) ?
- Si oui, impacte-t'elle la protéine codée par ce gène ?
- Entraîne-t-elle l'apparition d'un codon stop dans la séquence protéique (protéine tronquée par exemple) ?
- …
Être porteur d'une variation classée pathogène ne suffit pas pour prédire ou diagnostiquer une maladie. Par exemple, dans le cas des maladies récessives (mucoviscidose, drépnaocytose…), il faut que les deux copies du gène soient touchées pour être malade. Dans le cas des maladies à pénétrance incomplète (hémochromatose), vous pouvez être porteur de la mutation sans présenter un seul signe de la maladie.
Y a-t-il des variations pathogènes dans ces tests ?
Pour répondre à cette question, j'ai récupéré depuis la base de données ClinVar les variations pathogènes connues de classe 5 et j'ai fait l'intersection avec les 700 000 SNV de la puce illumina OmniExpress 24 utilisée par MyHeritage. Un notebook Python est disponible ici. Il en ressort une centaine de SNV classés pathogènes :
| Pathologie | rsid |
|---|
Parmi ces maladies génétiques, je me contenterai de commenter deux d'entre elles.
La mucoviscidose
22 SNV présents sont identifiés sur cette puce comme pathogènes pour la mucoviscidose. Il s'agit d'une maladie récessive très fréquente impliquant le gène CFTR situé sur le chromosome 7. Cela signifie que les deux copies du gène doivent êtres touchées pour entraîner la maladie. Une personne hétérozygote est porteuse saine et a un risque d'1 sur 2 de transmettre la variation à son enfant. On trouve dans la liste la variation rs75961395 ou VG07S29458, correspondant à la mutation c.254G>A Gly85Glu décrite dans la base de donnée CFTR-France. Elle fait partie des 30 mutations les plus fréquentes que l'on recherche systématiquement en routine (lors du diagnostic). Mais elle représente moins de 1 % des mutations chez les patients, loin derrière la mutation DF508, la plus connue et la plus fréquente.
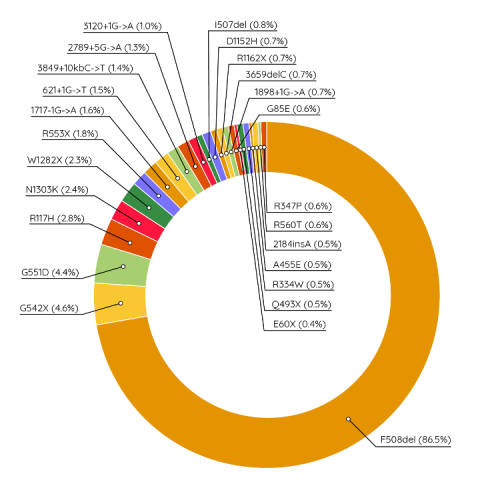
Source: Cystic Fibrosis Foundation Patient Registry 2014.
Prédisposition au cancer
S’il y a bien des tests génétiques extrêmement contrôlés, ce sont les tests d'oncogénétique indiquant la prédisposition au cancer avec une forte pénétrance. En étant porteur de ce type de variation pathogène, il y a une forte probabilité de développer un cancer. La liste de ces gènes est détaillée sur la page de l'INCa que je vous conseille de lire.
On y trouve le syndrome héréditaire de prédisposition au cancer du sein et de l'ovaire, médiatisé par l'actrice Angelina Jolie, probablement porteuse d'une mutation dans le gène BRCA1 et qui a eu recours à la chirurgie prophylactique.
Sur la puce, j'ai trouvé la variation rs28897743 ou i5009343 située sur le gène BRCA2. Cette variation est identifiée comme probablement pathogène. Je n'ai pas trouvé de papier sur cette mutation. Il y a également d'autres variations dans les exons mais non classées.
Pour les hommes, il y a aussi la variation rs721048, associée au cancer de la prostate qui, d'après ce papier, est fortement impliquée.
En conclusion
Parmi ces 700 000 SNV testés, très peu sont répertoriés dans ClinVar comme étant pathogènes. Pour certains syndromes à transmission dominante, vous seriez déjà au courrant si vous étiez atteint. Cependant, gardez en tête que les bases de données génétiques sont très loin d'être exhaustives. Chaque jour, de nouvelles variations sont découvertes comme pouvant être impliquées dans une maladie. C'est d'ailleurs pour cela que ces puces sont très utiles à la recherche. Vous n'êtes donc pas à l'abri que, dans 10 ans, ces données révèlent une information importante sur votre santé.
Je pense en particulier au score de risque polygénique associant la présence de plusieurs SNV à une maladie. On trouve déjà sur Internet ce genre de test pour la maladie d'Alzheimer par exemple. Bref, tous ces tests sont, à mon sens, éthiquement borderline. Surtout quand de la publicité est faite sur YouTube sachant que sa dirigeante Susan Wojcicki est la sœur de Anne Wojcicki, elle-même dirigeante de… 23andMe.
Je vous conseille donc de réfléchir à deux fois avant de faire ce genre de test, de bien lire le consentement et de le faire de façon anonyme. Il faut par ailleurs garder en tête qu'il n'y a pas plus identifiant que notre propre génome et que cet anonymat est relatif. Je n'en ai pas parlé, mais vous n'êtes pas l'abri que ces données arriveront un jour entre de mauvaises mains. L'argument c'est ultra sécurisé on l'a déjà entendu avec tous les services web qui se sont ensuite fait pirater.
Je tiens enfin à recommander à toutes celles et ceux qui ont réalisé le test d'éviter de s'essayer à l'autodiagnostic. Si vous avez la moindre inquiétude quant à vos résultats, consultez un médecin généticien en demandant à votre médecin généraliste.
Références
- Règles de bonne pratique génétique
- ACMG
- États généraux de bioéthique
- Prédisposition oncogénétique - INCa
Merci pour la relecture
- Charlotte Andrieu
- @Oodnadatta
Ce site est versionné sur GitHub. Vous pouvez corriger des erreurs en vous rendant à cette adresse
Go Top