Un filtre de bloom est une structure de donnée permettant de savoir si un élément est présent ou non dans une liste. Cette structure est très efficace d'un point de vue mémoire lorsque vous travaillez sur de grande liste. En python, l'utilisation d'un filtre de Bloom peut s'écrire :
L = ["mario","zelda","daisy"]
has_item(L,"sonic") # Return False
has_item(L,"mario") # Return True
Le revers de la médaille, c'est qu'avec cette méthode il existe des faux positifs, c'est à dire que la fonction renvoie Vrai alors que le mot n'est pas dans la liste... Inutile vous me direz ? Non, car dans certain cas, on se fiche des faux positifs. Par exemple, on peut être amené à savoir si un mot est bien absent de la liste. Et dans ce cas, c'est tout à fait possible avec les filtres de Bloom, car il n'y a pas de faux négatifs!
Dans d'autre cas, notamment dans l'analyse des Big data, les performances d'un algorithme sont prioritaires au risque de faux positifs. On pourra alors paramétrer l'algorithme de façon à minimiser le nombre de faux positifs.
Fonction de hachage
Les filtres de Bloom utilisent des fonctions de hachage. Une fonction de hachage est une fonction qui, à partir d'une entrée renvoie une "empreinte" ou encore une "signature" permettant d’identifier l'entrée. Les fonctions de hachage sont utilisées dans de nombreux cas, notamment en cryptographie ou dans les structures de données de type dictionnaire.
Dans le cas des filtres de Bloom, une fonction de hachage renvoie un unique entier compris entre 0 et n, choisi de façon uniforme. On peut créer autant de fonction de hachage qu'on le désire.
# Une fonction de hachage
hash1("mario") # Return 3
hash1("zelda") # Return 4
hash1("daisy") # Return 3 .. Collision
# Une autre fonction de hachage
hash2("mario") # Return 7
hash2("zelda") # Return 5
hash2("zelda") # Return 3
# Encore une autre ...
hash3("mario") # Return 54
Attention tout de même! Avec des fonctions de hachage il peut se produire des collisions, c'est-à-dire que pour deux entrées différentes il y a un même hash. En jouant avant la taille de n et connaissant les entrées, on peut minimiser la probabilité de collision.
Utilisation du filtre de bloom
L’utilisation d'un filtre de Bloom comprend 2 étapes. La première consiste à hacher tous les éléments de notre liste et les "installer" dans un vecteur booléen de taille n en utilisant k fonctions de hachage différentes.
La deuxième, teste la présence d'un élément en recherchant son hash dans ce vecteur booléen .
Création du vecteur booléen
Choisissons pour l'exemple, un vecteur de taille n=10, et initialisons le avec des zéros. Puis choissons k=3 fonctions de hachages différentes, que nous notons h0,h1 et h2. Les hashs obtenus correspondent à une position dans le vecteur. Les valeurs possibles des hashs doivent être alors comprises entre 0 et 9.

Enfin, commençons par installer notre liste, avec le première élément "mario", comme l'illustre la figure suivante :
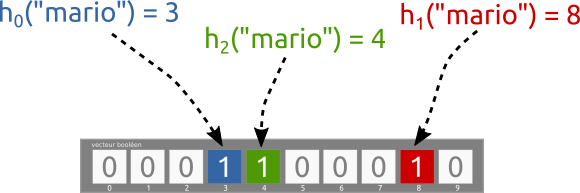
Ajoutons maintenant le mot "zelda" :
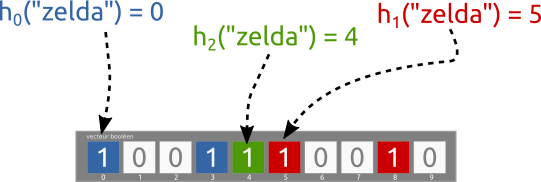
Avec le mot "zelda", il y a eu collision avec la fonction de hashage h2. C'est ce qui est l'origine des faux positifs.
Test de présence
Si vous avez compris jusque là, vous devriez comprendre comment tester la présence du mot "sonic". Si le mot a été "installé" , alors nous devrions retrouver tous les hashs obtenus par les 3 fonctions de hachages sur le mot "sonic". Ce qui n'est pas le cas ici, comme l'illustre la figure suivante. Le mot sonic n'est pas présent dans la liste.

Estimation des faux positifs
Avec cette algorithme, il n'y a jamais de faux négatifs, mais des faux positifs. C'est-à-dire le fait de répondre qu'un élément est présent alors que non.
Pour compenser ce problème, nous pouvons créer le filtre de façon à minimiser le nombre de faux positifs via les 3 paramètres suivants :
- n = La taille du vecteur booléen (10)
- m = Le nombre d'élément d'une liste (3)
- k = Le nombre de fonction de hachage (3)
Considérons une position j dans notre vecteur. La probabilité qu'une fonction de hachage fasse passer la valeur de j de 0 à 1 est de $\frac{1}{n}$. L'inverse, c'est-à-dire la probabilité que la valeur de j ne change pas, est donc de $1-\frac{1}{n}$.
Après avoir rempli le vecteur booléen, avec m élément et k fonction de hachage, la probabilité de ne pas changer j est donc de $(1-\frac{1}{n})^{km}$.
Cette équation peut se réduire en considérant l'égalité approximative suivante:
$(1-\frac{1}{n})^n\approx \frac{1}{e} = e^{-1}$
L'équation précédente peut se réduire alors :
$(1-\frac{1}{n})^{km} \approx e^{\frac{-km}{n}}$
Au final, la probabilité d'avoir des faux positifs équivaut à la probabilité d'avoir toutes les positions du vecteur booléen à 1, pour les k fonctions de hachage. On obtient ainsi la formule finale suivante :
$P_{faux-positif} = ( 1 - e^{\frac{-km}{n}})^k$
Application
Sachant que nous avons une liste de m élément, quelles sont les valeurs de k et n que nous pouvons choisir pour atteindre une probabilité p de faux positifs ? En faisant un peu d'algèbre, la meilleure valeur de k est : k = $ln(2)\frac{n}{m}$
Et le rapport suivant doit être satisfait: $\frac{n}{m} = 0.7ln(\frac{1}{p})$.
Par exemple, pour atteindre une probabilité $p<\frac{1}{1000}$, il suffit de choisir $\frac{n}{m} > 0.7ln(1000) \approx 7$. Avec m = 1000 éléments on choisira donc n = 7000 et k = 5.
Pour plus d'information sur la démonstration mathématique, regardez la vidéo youtube en référence.
Conclusion
Avec toutes ces explications, vous pouvez je pense, réaliser votre propre filtre de Bloom. Utiliser la librairie cityhash pour les fonctions de hachage, ça devrait marcher. Mais si vous avez la flemme, j'ai vu qu'il existait une librairie toute faite pybloom. L'article sur le blog de Max Burstein est plus orienté code et explique pas à pas la création en python d'une class BloomFilter.
Enfin, pour les bioinformaticiens, je vous invite à lire cette article : Efficient counting of k-mers in DNA sequences using a bloom filter.
Référence
- Youtube Bloom Filter (anglais)
- bioinfo-fr
- Create a simple bloom filter
- Efficient counting of k-mers in DNA sequences using a bloom filter.
Ce site est versionné sur GitHub. Vous pouvez corriger des erreurs en vous rendant à cette adresse
Go Top