Cela fait un bon moment que j'entends parler des fichiers Parquet. Un format binaire beaucoup plus léger
que les fichiers CSV pour le stockage des tableaux de données.
Lorsque j'ai vu pour la première fois un collègue faire une requête SQL quasi instantanée sur des millions des lignes répartis sur différents fichiers parquets, je me suis dit les yeux grands écarquillés, que c'était peut être un peu plus que ça.
En effet, aujourd'hui le format parquet est utilisé en big data pour stocker et interroger de façon efficace des données
volumineuses grâce à un modèle orienté colonne basé sur Apache Arrow que je décrirai juste après.
À cela, s'ajoutent de nouveaux outils en Python pour pouvoir manipuler et interroger ces fichiers.
Dans ce billet de blog, je vous propose d'utiliser pola.rs et duckDB, pour explorer les données d'un fichier VCF volumineux provenant de 1000genomes.
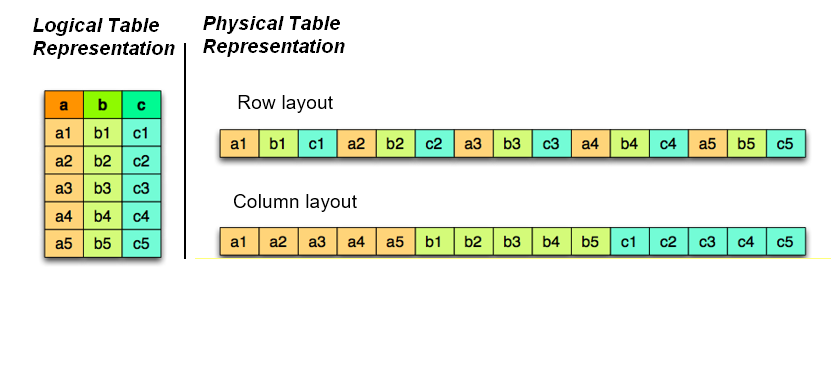
Architecture d'un fichier parquet
Base de données orientée colonnes
Les bases de données classiques ( MYSQL, SQLite, Oracle... ) sont des architectures orientées en ligne. C'est-à-dire que les lignes d'une table sont sauvegardées de manière contiguë en mémoire. Cela permet d'insérer ou de supprimer facilement des enregistrements. Revers de la médaille, il est plus coûteux de faire du calcul sur une colonne entière, car cela nécessite de parcourir l'ensemble des lignes. Ces bases de données sont optimisées pour le transactionnel et sont utilisées pour des systèmes OLTP (OnLine Transactional Processing), par exemple une base de données de production gérant des utilisateurs.
Dans un fichier parquet, ce sont les colonnes qui sont sauvegardées de manière contiguë en mémoire. Ceci permet de faire
des opérations de façon très efficace sur les colonnes au détriment des opérations transactionnelles. Cette architecture est très performante pour des systèmes OLAP (OnLine Analytical Processing). Par exemple un entrepôt de données destiné à être lu uniquement.
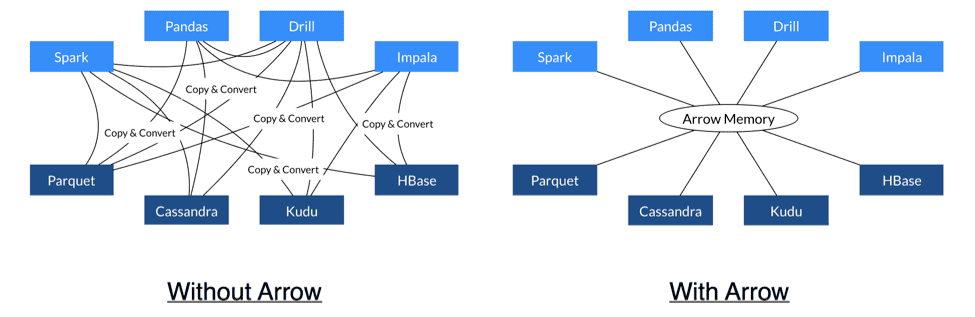
Apache Arrow
Apache Arrow est un format standard de donnée orienté colonne pour la mémoire vive. C'est-à-dire qu'il décrit, indépendamment du langage de programmation,comment représenter un tableau dans votre RAM. Par exemple, si vous manipulez les mêmes données stockées dans un DataFrame Python ou un DataFrame R, la structure mémoire sous-jacente sera la même. Autrement dit, vous allez pouvoir transférer un DataFrame Python vers un DataFrame R, sans faire la moindre copie ou transformation. Et lorsque l'on travaille avec beaucoup de données, cela est loin d'être négligeable.
Le format parquet développé par Apache, est entièrement compatible avec Arrow. La sérialisation et la déserialisation d'un DataFrame,
c'est à dire l'écriture et la lecture d'un fichier parquet sera très performante avec un minimum de transformation.
Du VCF au parquet avec polars
Pour lire et écrire des fichiers parquet avec Python, vous pouvez utiliser la libraire pandas. Cependant, pandas n'est pas basée sur Arrow et reste très lente pour manipuler de gros volumes de données. Nous utiliserons ici les performances quasi magiques de la libraire pola.rs pour transformer un fichier VCF en fichier parquet.
Pola.rs est écrit en Rust, est compatible avec Arrow, supporte nativement le multithreading et propose une Lazy évaluation des transformations. Je vous invite à jeter un œil sur la documentation Python pour vous familiariser avec l'API qui diffère de celle de pandas.
Téléchargement du fichier VCF
Téléchargeons un fichier VCF provenant du projet 1000Genomes et décompressez-le. Nous allons lire le fichier VCF comme un fichier CSV en mode Lazy. Ce dernier mode ne fonctionne pas encore pour les fichiers compressés.
# Téléchargement
wget ftp://ftp.1000genomes.ebi.ac.uk/vol1/ftp/data_collections/1000_genomes_project/release/20190312_biallelic_SNV_and_INDEL/ALL.wgs.shapeit2_integrated_snvindels_v2a.GRCh38.27022019.sites.vcf.gz
# Decompression
gzip -d ALL.wgs.shapeit2_integrated_snvindels_v2a.GRCh38.27022019.sites.vcf.gz
Création du fichier parquet avec pola.rs
Après avoir installé pola.rs via:
pip install pola.rs
- Lisez le fichier VCF comme un CSV avec pl.scan_csv
- Sélectionner les colonnes CHROM, POS, REF, ALT
- Écrire le fichier variants.parquet avec sink_parquet
import pola.rs
pl.scan_csv(
"ALL.wgs.shapeit2_integrated_snvindels_v2a.GRCh38.27022019.sites.vcf",
skip_rows=40, # Je saute les 40 premieres lignes de commentaires
sep="\t", # Séparateur TSV
dtypes={"#CHROM": pl.Utf8}, # Je précise le type, sinon la colonne est considérée comme un int
).select([ # Je sélectionne les colonnes souhaitées
pl.col("#CHROM").alias("CHROM"), # Je renomme ici la colonne avec alias
pl.col("POS"),
pl.col("REF"),
pl.col("ALT")]
).sink_parquet( # Écriture du fichier parquet
"variants.parquet"
)
Je vous conseille de regarder le temps d'exécution et faire un htop pour voir la parallélisation opérée ainsi que la consommation mémoire. C'est assez bluffant. Je mets moins de 5 secondes sur mon ordinateurs perso (AMD Ryzen 9 5900X) pour traiter 78'229'218 variants. Et pour la consommation mémoire, Les fonctions scan_csv et sink_csv permettent de faire la transformation du VCF sans le charger en mémoire. Regardez aussi les tailles du fichier. 225Mo pour le fichier parquet et 1.3Go pour son équivalent en CSV. En effet, les fichiers parquets sont compressés naturellement du fait du modèle orienté colonne.
Requête SQL avec DuckDB
À présent essayez de requêter sur ce fichier. Nous pourrions le faire avec pola.rs, mais nous allons plutôt faire une requête SQL en utilisant duckDB qui s'installe tout aussi facilement avec la commande suivante :
pip install duckdb
Pour exécuter une requête SQL sur un fichier parquet, il suffit de considérer le fichier comme le nom d'une table SQL :
# Simple requête pour visualiser le contenu du fichier parquet
duckdb.sql("SELECT * FROM 'variants.parquet'")
# ┌─────────┬─────────┬─────────┬─────────┬─────────┐
# │ #CHROM │ POS │ REF │ ALT │ ID │
# │ varchar │ int64 │ varchar │ varchar │ varchar │
# ├─────────┼─────────┼─────────┼─────────┼─────────┤
# │ 1 │ 10416 │ CCCTAA │ C │ . │
# │ 1 │ 16103 │ T │ G │ . │
# │ 1 │ 17496 │ AC │ A │ . │
# │ 1 │ 51479 │ T │ A │ . │
# │ 1 │ 51898 │ C │ A │ . │
# │ 1 │ 51928 │ G │ A │ . │
À présent, essayons de faire plus compliqué en comptant le nombre de transitions et de transversions. C'est à dire, le nombre de combinaisons A>T, C>G etc ...
# A partir des SNPS len(ref)=1 et len(alt)=1
# Je construis une liste [ref, alt] que je trie
# Je fait un groupby et un comptage
q = """
"SELECT list_sort([ref,alt]) AS mut, COUNT(*) as count FROM 'variants.parquet'
WHERE len(ref) = 1 AND len(alt)=1 GROUP BY mut
"""
duckdb.sql(q)
# ┌───────────┬──────────────┐
# │ mut │ count │
# │ varchar[] │ int64 │
# ├───────────┼──────────────┤
# │ [C, T] │ 24782079 │
# │ [A, G] │ 24828822 │
# │ [G, T] │ 6103978 │
# │ [A, T] │ 5140035 │
# │ [A, C] │ 6086989 │
# │ [C, G] │ 6315729 │
# └───────────┴──────────────┘
Vous devriez retrouver après quelques secondes les mêmes proportions que j'ai déjà détaillées dans un précédent billet.
Autres astuces
Le partitionnement
Niveau performance, c'est déjà bluffant. Mais il existe différentes méthodes d'optimisation pour être plus performant suivant l'usage des données. Le partitionnement consiste à découper votre fichier parquet en plusieurs fichiers parquet depuis une ou plusieurs colonnes. Par exemple, je peux partitionner le fichier parquet variants.parquet par chromosomes. Si je dois chercher un variant sur le chromosome 8, je peux regarder uniquement dans le fichier correspondant. Inutile de parcourir les variants du chromosomes 2.
Construisons une partition sur la colonne chromosome avec duckDB :
duckdb.sql(
"COPY (SELECT * FROM 'variants.parquet') TO 'chromosomes' (FORMAT PARQUET, PARTITION_BY (CHROM))"
)
Après avoir exécuté cette requête, vous devriez avoir un dossier chromosomes contenant de
nombreux fichiers triés par chromosomes.
Pour sélectionner vos variants depuis ce dossier, il suffit d'utiliser le caractère étoile ou des expressions régulières pour sélectionner les sources de données souhaitées.
Dans l'exemple suivant, je sélectionne tous les variants à partir de tous les fichiers :
duckdb.sql("SELECT * FROM 'chromosomes/*/*.parquet'")
Combiner Pola.rs et duckdb
Une dernière astuce pour la fin. Pola.rs et duckdb sont de très bon amies et sont interchangeables. Vous pouvez switcher de l'un à l'autre très facilement ( Merci Arrow ).
# Passer de duckdb à pola.rs
df = duckdb.sql("SELECT * FROM 'variants.parquet' WHERE CHROM='22'").pl()
# Passer de pola.rs à Duckdb
df = pl.read_csv(....)
duckdb.sql("SELECT * FROM df")
Conclusion
Pola.rs et Duckdb sont des technologies nées du big data qui, je vous parie, vont devenir des références pour la manipulation des données volumineuses et remplacer leurs prédécesseurs comme Pandas.
Références
- Pola.rs
- Duckdb
- Apache Parquet pour le stockage de données volumineuse
- Demystifying the Parquet file Format
- Clickhouse : une autre base OLAP
Ce site est versionné sur GitHub. Vous pouvez corriger des erreurs en vous rendant à cette adresse
Go Top